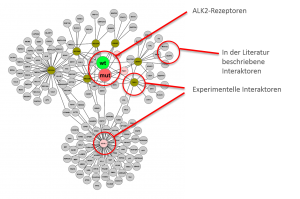
Untersuchung des BMP-Signalwegs in HEK293-Zellen
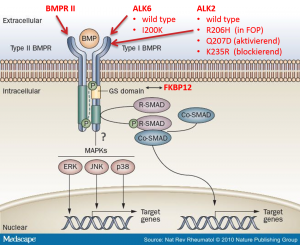
Folgende relevante Proteine wurden für die Analyse ausgewählt, da sie Schlüsselkomponenten in der frühen Signalkaskade von BMP darstellen:
- ALK2 wildtype receptor
- ALK2 mutated receptors:
- R206H (causes FOP (Fibrodysplasia ossificans progressiva))
- Q207D (constitutive active)
- K235R (dominant negative)
- ALK6 wildtype receptor
- ALK6 mutated receptor (I200K): causes brachydactyl (shortness of the fingers and toes)
- BMPRII: type II receptor
- FKBP12
Die Schlüsselproteine sind auf der folgenden Abbildung nochmals im Kontext des Signalwegs gezeigt.
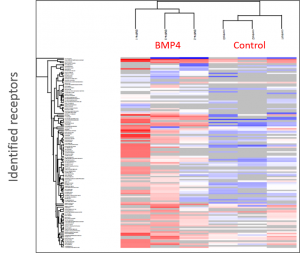
Identifikation der BMP-Rezeptoren und Signalproteine
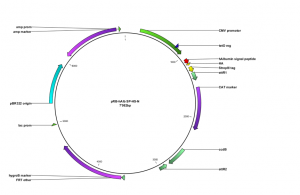
Konstruktion neuer Expressionsvektoren
Zunächst mussten verschiedene Vektoren hergestellt werden, um die verschiedenen Schlüsselproteine exprimieren zu können.
Untenstehend ist der Basis-Vektor gezeigt. Es handelt sich um einen eukaryotischen Expressionsvektor, welcher folgende Komponenten trägt:
- Strep II und HA Epitop-Tags zur Aufreinigung und Detektion der Schlüsselproteine
- Induzierbarer Promoter zur regulierten Expression
- Verschiedene Selektionsmarker zur Erzeugung stabiler Zelllinien
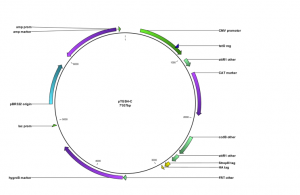
Konstruktion eines neuen Expressionsvektors für ALK2
Zusätzlich wurde ein spezieller Expressionsvektor für die Expression der BMP-Rezeptoren konstruiert.
Dies ist notwendig, da die BMP-Rezeptoren N-terminal ein spezielles Element tragen, welches bei der Expression aus konventionellen Vektoren verloren gehen würde. Der modifizierte Expressionvektor sorgt für die korrekte Expression dieses Elements.
Klonierung der Konstrukte
Anschliessend wurden die untenstehenden Konstrukte generiert, indem die für die Schlüsselproteine kodierenden Gene in die Expressionsvektoren kloniert wurden:

Transfektion und Etablierung stabiler HEK293-Linien
- HEK293-Zellen wurden mittels der Calciumphosphat-Methode transfektiert
- Die folgenden Konstrukte wurde hierfür eingesetzt:
- ALK2wt (wildtyp)
- ALK2 Q207D
- ALK2 K236R
- ALK2 R206H
- Mittels Selektion wurden stabile Linien erzeugt, welche die Proteine exprimieren
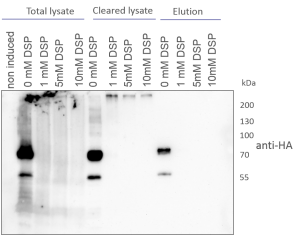
- Der Nachweis der Expression erfolgte mittels Proteinextraktion aus den Zellen, gefolgt von Auftrennung mittels SDS-PAGE und Western Blotting mit einem antiHA-Antikörper
- Anschliessend wurde das Extraktions-/Aufreinigungsprotokoll optimiert, um die Sensitivität zu optimieren
Etablierung der HEK293-Linien und Optimierung der Aufreinigung
- Generierung von 8 Zelllinien, welche die Schlüsselproteine exprimieren
- Optimierung der Aufreinigung, um möglichst viele Interaktionen sichtbar zu machen

Etablierung der HEK293-Linien und Optimierung der Aufreinigung
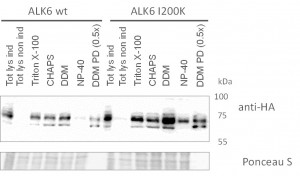
Etablierung der HEK293-Linien und Optimierung der Aufreinigung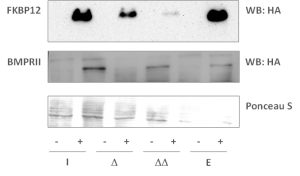
Test der Funktionalität des Signalwegs
- Die Aktivität des BMP-Signalwegs kann mittels eines «Indikator-Proteins» getestet werden
- Der ALK2-Rezeptor hat eine intrazelluläre Kinase-Domäne
- Wird ALK2 durch Stimulation mit dem Liganden BMP4 aktiviert, so wird die Kinase-Domäne aktiv und modifiziert das Signalprotein Smad
- Die Aktivierung erfolgt via Modifikation mit einem Phosphat-Rest (Phosphorylierung)
- Die Phosphorylierung von Smad kann mittels Western Blotting mit einem anti P-Smad Antikörper nachgewiesen werden
Resultate
Die Schlüsselproteine sind exprimiert
Der Signalweg kann stimuliert werden und die Phosphorylierung von Smad 1, 5 und 8 ist detektierbar

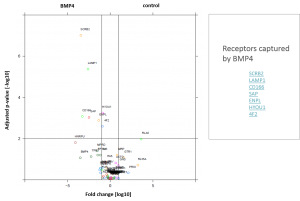
Aufreinigung und Identifikation der Proteinkomplexe
- Die Proteinkomplexe aus den stabilen HEK293-Linien wurden mittels des optimierten CaptiVate Pulldown/MS-Verfahrens aufgereinigt
- Für jede Linie wurden zunächst mehrere Pilot-Aufreinigungen durchgeführt und getestet
- Sodann wurden Aufreinigungen im Triplikat mit grossen Zellmengen durchgeführt
- Die Zellen wurden aufgebrochen und die Proteine extrahiert
- Die Aufreinigung geschah mittels eines Strep-Säulenmaterials
- Die aufgereinigten Proben wurden für die Massenspektrometrie vorbereitet, durch Nano-LC aufgetrennt und in der LTQ Orbitrap analysiert
- Die Datensätze wurden mittels verschiedenen bioinformatischen Methoden aufbereitet und analysiert
- Ergebnis ist eine Liste von Interaktoren des Bait-Proteins
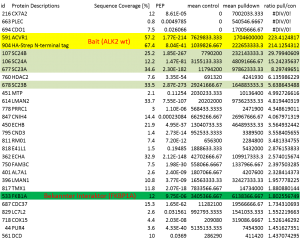
Zusammenfassung der in Hek293-Zellen durchgeführten Pulldown-Experimente
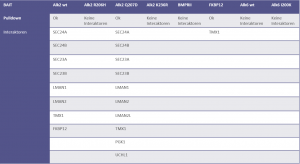
Resultate
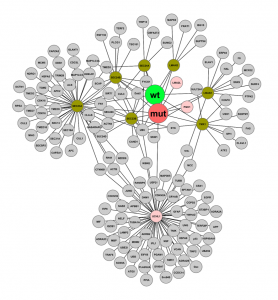
- Für ALK2 wildtype konnten mehrere Interaktoren identifiziert werden
- Für die ALK2-Mutante Q207D wurden ebenfalls Interaktoren gefunden, welche stark mit den für ALK2 wildtype gefundenen Interaktoren überlappen
- Zusätzlich wurden für ALK2 Q207D weitere Interaktoren identifiziert, während für ALK2 R206H und ALK2 K236R keine Interaktoren gefunden wurden
- Der BMPRII Rezeptor lieferte ebenfalls keine Interaktoren, was an der sehr schwachen Expression des Rezeptors liegen könnte
- Für das Modulator-Protein FKBP12 konnte ein Interaktor identifiziert werden, ebenfalls wurde FKBP12 als Interaktor von ALK2 wildtype gefunden
- Pulldowns mit den beiden ALK6-Rezeptoren lieferten keine Interaktoren
- Die mit den ALK2-Rezeptoren und FKBP12 identifizierten Interaktoren erlauben die Abbildung des Rezeptor-proximalen Signalweges, sowie der Sorting- und Degradationsmaschinerie, welche den Abbau des ALK2-Rezeptors steuert